Hochdurchsatzsequenzierung (Next-Generation Sequencing, NGS) Einheit
Die Hochdurchsatzsequenzierung (Next-Generation Sequencing, NGS) Einheit, ist ein integrierte Einheit des BNITM, die NGS-Unterstützung bietet und gehört bei vielen Forschungsprojekten am BNITM mittlerweile zum festen Bestandteil des Methodenumfangs und wird routinemäßige genutzt. Die Einheit betreibt mehrere Next-Generation-Sequenzer, die die Sequenzierung durch Synthese-Technologie von Illumina verwenden, darunter NextSeq2000, NextSeq550, MiSeq und iSeq100. Sie unterhält ein voll ausgestattetes Labor und qualifiziertes Personal, das eine Vielzahl von Sequenzierungsanwendungen von der Probenpräparation bis zur Sequenzierung durchführen kann. Die Einheit unterstützt dabei aktiv die Planung und Durchführung der Projekte. Wir haben verschiedene Arbeitsabläufe und Pipelines für die Probenpräparation etabliert, um den Durchsatz zu erhöhen und technische Variationen zu minimieren. Durch die Zentralisierung von Geräten und Fachwissen haben wir die Effizienz und Qualität der generierten Daten erhöht. Die Vorbereitung von Bibliotheken für verschiedene Anwendungen wird von einer robusten Instrumenteninfrastruktur und einem geschlossenen Laborsystem unterstützt (z. B. Covaris, Bioanalyzer, Qubit, Pippin Prep, qPCR und mehr). Die NGS-Einheit bietet verschiedene Analysen im Single- oder Paired-End-Sequenziermodus an, einschließlich Multiplexing-Bibliotheken.
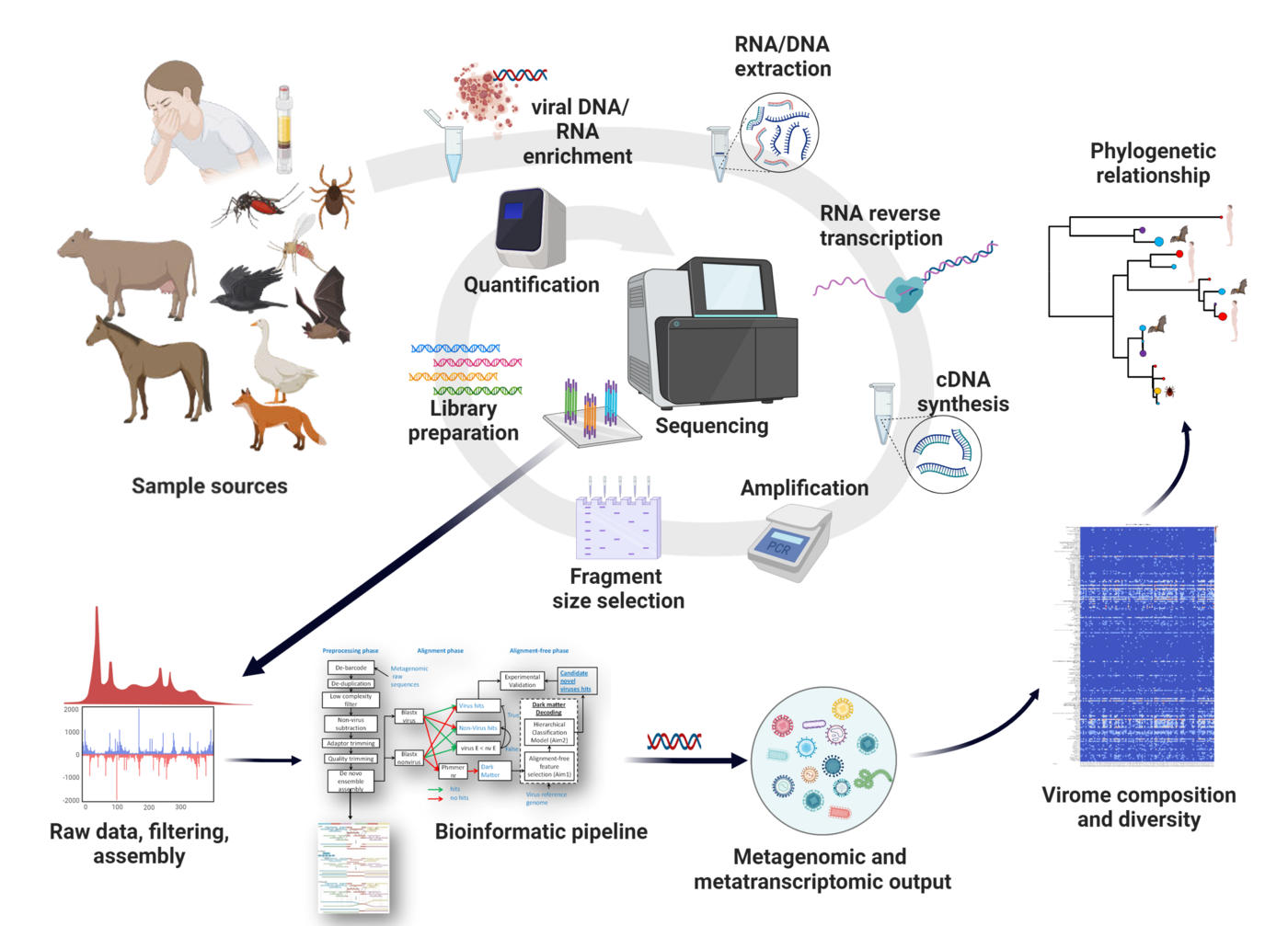

Das Methodenportfolio beinhaltet standardisierte Workflows und Pipelines für verschiedene NGS-Anwendungen:
- Virale Metagenomik und Metatranskriptomik: RNA-Seq/DNA-Seq
- Vollständige Transkriptom-Sequenzierung: Total RNA-Seq, mRNA
- Genomsequenzierung von Insekten
- Exom-Sequenzierung
- Entdeckung kleiner nicht-kodierender RNAs: ncRNA-Seq.
- Genomweite DNA-Methylierungsanalyse: Methyl-Seq.
- De-novo-Sequenzierung und Re-Sequenzierung von genomischer DNA.
- Gezielte Anreicherung und Sequenzierung (Sequence Capture).
- Einzellige genomische Anwendungen (scRNA-Seq, scDNA-Seq)
- 16S Metagenomische Sequenzierung
- miRNA- und kleine RNA-Analyse
- Langstrecken-Amplikon-Sequenzierung














