Neue Nachweismethode nutzt Blutspenden als Frühwarnsystem für das West-Nil-Virus
Forschende des Bernhard-Nocht-Instituts für Tropenmedizin (BNITM) haben eine neue Sequenziermethode entwickelt, die das West-Nil-Virus in Blutspenden nachweisen kann. Die hochsensitive Methode macht es möglich, selbst winzige Virusmengen aufzuspüren und deren Erbgut vollständig zu analysieren. Die Forschenden können so Virusvarianten frühzeitig erkennen und deren Ausbreitung in Deutschland verfolgen. Die Forschenden zeigten zudem: Das Virus wurde mehrfach unabhängig aus Zentraleuropa nach Deutschland eingetragen und hat sich inzwischen in mehreren östlichen Bundesländern etabliert. Die Studie ist kürzlich im Journal of Infection erschienen.
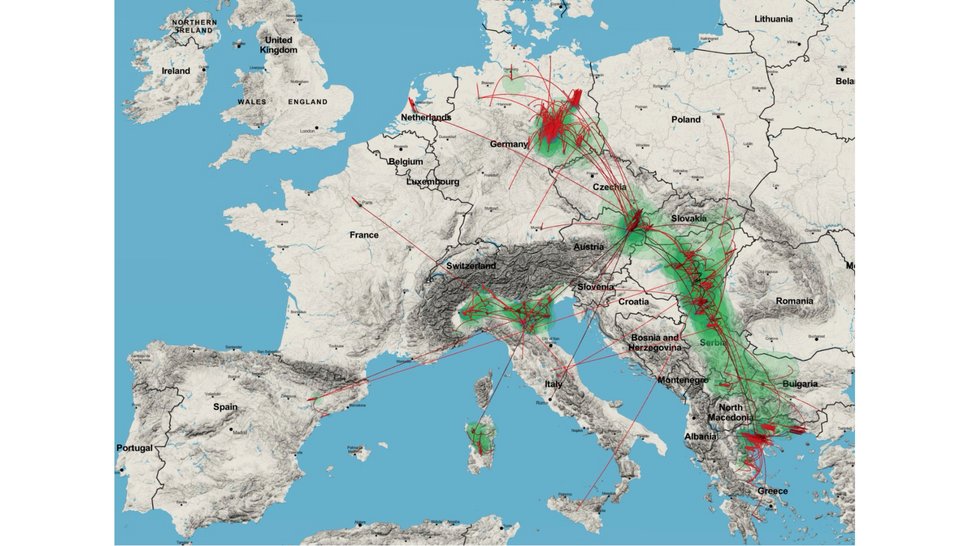
Stechmücken übertragen das West-Nil-Virus (WNV) , das beim Menschen in seltenen Fällen schwere neurologische Erkrankungen verursachen kann. Seit seiner Entdeckung in Deutschland im Jahr 2018 ist das Virus in mehreren östlichen Bundesländern verbreitet. Um Infektionsketten und Virusvarianten zu erkennen, ist eine genaue genetische Überwachung (Genomüberwachung) entscheidend.
„Je früher wir das Virus nachweisen, desto schneller können Gesundheitsbehörden reagieren und Ausbrüche verhindern“, sagt Dr. Dániel Cadar, Leiter der Arbeitsgruppe Virus Metagenomik und Evolution am BNITM und Letztautor der Publikation. „Genomische Überwachung ist unser Frühwarnsystem. Sie zeigt uns auch, wann und wo sich neue Virusvarianten etablieren.“
Neue Methode erfasst selbst winzige Virusmengen
Das Team um Cadar hat zusammen mit Kooperationspartner:innen anderer Institute ein neues, sehr sensitives Verfahren für den Nachweis von West-Nil-Virus-Erbgut entwickelt. Mit diesem sogenannten Amplikon-basierten Sequenzieransatz können die Forschenden winzige Mengen an Virus-Erbgut zuverlässig nachweisen und vollständig genetisch analysieren. Bisher wurden zum Nachweis metagenomische Verfahren genutzt, die das gesamte genetische Material einer Probe auslesen und dabei sämtliche DNA- und RNA-Fragmente von Viren, Bakterien und Zellen gleichzeitig erfassen. Bei sehr geringen Virusmengen stößt diese Methode aber an ihre Grenzen und sie kann das Virus nicht mehr zuverlässig nachweisen.
Das neue Verfahren basiert auf gezielten „Verstärkungsreaktionen“ (Amplikons), die spezifische Abschnitte des Virus-Erbguts vervielfältigen. Dadurch können die Wissenschaftler:innen auch Proben mit sehr niedriger Viruslast erfolgreich analysieren.
„Unsere Methode ermöglicht die Genomsequenzierung auch dann, wenn nur wenige Viruspartikel in der Probe vorhanden sind. Das war bisher kaum möglich. Im Vergleich zu metagenomischen Verfahren ist unsere Methode bis zu hundertmal empfindlicher“, sagt Cadar. „Damit schaffen wir die Grundlage für eine schnelle, präzise und kosteneffiziente Virusüberwachung.“


Blutspenden als Frühwarnsystem
Die neue Sequenziermethode ermöglicht es nun erstmals, Blutspenden mit sehr geringen Virusmengen erfolgreich auszuwerten. Deshalb arbeiteten die BNITM-Forschenden für die Studie eng mit dem Deutschen Roten Kreuz zusammen.Die Blutspendeproben sind besonders wertvoll, weil sie flächendeckend, regelmäßig und unabhängig von Krankheitssymptomen gesammelt werden. So entsteht ein einzigartiges Bild der Viruszirkulation in der Bevölkerung. Erstautor Gábor Endre Tóth erklärt:
„Blutspenden sind ideale Frühwarnsysteme. Sie geben uns ein genaues Bild, wo und wann das Virus im Umlauf ist, oft noch bevor klinische Fälle auftreten. Sie verbinden routinemäßige Gesundheitsvorsorge mit epidemiologischer Überwachung. Dank dieser Daten können wir Infektionsketten nachvollziehen und die Ausbreitung des Virus viel besser verstehen.“
Auf den Pfaden des Virus
Die Forschenden analysierten 43 für das West-Nil-Virus positive Blutspenden aus den Jahren 2020 bis 2024 genetisch. Das waren so viele wie nie zuvor in Deutschland. Die Forschenden „verfolgten“ das Virus genetisch, da heißt, sie konnten nachvollziehen, woher ein Virus stammt, wie es sich verändert hat und welche Varianten aktuell im Umlauf sind. Die genetischen Analysen zeigen: Das West-Nil-Virus wurde mehrfach unabhängig aus Zentraleuropa nach Deutschland eingebracht. Es handelt sich also nicht um ein einzelnes Ereignis, sondern das Virus gelangte über verschiedene Wege und zu unterschiedlichen Zeitpunkten nach Deutschland. Mit der neuen Methode ließ sich außerdem erkennen, dass sich sechs unterschiedliche Virus-Untergruppen („Subkladen“) der WNV-Linie 2 gebildet haben. Diese Varianten haben sich in mehreren Regionen Deutschlands etabliert, vor allem in Berlin, Brandenburg, Sachsen und Sachsen-Anhalt.
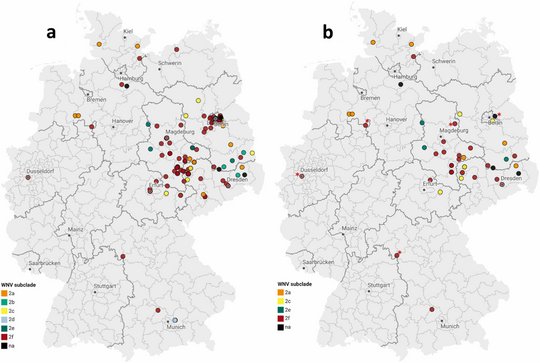
„Unsere Studie liefert den bislang umfassendsten Überblick über die genetische Vielfalt und regionale Verbreitung des West-Nil-Virus in Deutschland. Das Virus ist längst bei uns heimisch geworden und hat sich weiterentwickelt“, so Tóth. „Mit unserer Methode können wir nun präzise und in Echtzeit verfolgen, wie sich das Virus anpasst, wie es wandert und welche genetischen Linien sich durchsetzen.“
Grundlage für zukünftige Gesundheitsüberwachung
Die Studie belegt, dass die Kombination aus empfindlicher Sequenziertechnik und Blutspende-Überwachung ein effektives Instrument für die frühe Erkennung, Nachverfolgung und Bewertung von Virusausbrüchen ist. Dies gilt nicht nur für das West-Nil-Virus, sondern auch für andere durch Stechmücken übertragene Krankheitserreger.
„Dieses Verfahren stärkt die öffentliche Gesundheit in Deutschland erheblich“, sagt Cadar. „Wir können Virus-Einträge früh erkennen, ihre Entwicklung verfolgen und die regionale Ausbreitung besser verstehen. So können Gesundheitsbehörden schneller reagieren und Deutschland besser auf zukünftige Ausbrüche vorbereiten.“
Das am BNITM entwickelte Verfahren und die dazugehörige bioinformatische Pipeline sind im ergänzenden Material des Manuskriptes öffentlich zugänglich. Dadurch können künftig auch andere Labore in Europa die Methode anwenden, um Virusausbrüche frühzeitig zu erkennen. Die Forschenden betonen, wie wichtig eine kontinuierliche und vernetzte Überwachung Stechmücken- oder anderer Insekten-übertragener Krankheitserreger ist, denn ihre Verbreitung dürfte sich durch den Klimawandel weiter verstärken.
Originalpublikation:
Tóth, G.E. et al., Blood donors as sentinels for genomic surveillance of West Nile virus in Germany using a sensitive amplicon-based sequencing approach, Journal of Infection 2025, DOI: 10.1016/j.jinf.2025.106647
Funding:
Bundesministerium für Forschung, Technologie und Raumfahrt (BMFTR)
Bundesministerium für Landwirtschaft, Ernährung und Heimat (BMLEH)
Ansprechperson
Dr. Dániel Cadar
Arbeitsgruppenleiter
Telefon : +49 40 285380-840
Fax : +49 40 285380-400
E-Mail : cadar@bnitm.de
Dr. Anna Hein
Presse- & Öffentlichkeitsarbeit
Telefon : +49 40 285380-269
E-Mail : presse@bnitm.de
Weiterführende Informationen






