Neue Methode beschleunigt die Malaria-Forschung
Veröffentlichung in Nature Methods
Hamburg, 14. März 2017 – Der Malariaerreger Plasmodium falciparum ist schon seit mehr als hundert Jahren bekannt, dennoch ist die Funktion von mehr als der Hälfte seiner rund 5.500 Gene bis heute nicht geklärt. Mit einer neuen gentechnischen Methode können Wissenschaftlerinnen und Wissenschaftler des Bernhard-Nocht-Instituts für Tropenmedizin (BNITM) nun gezielt und schnell feststellen, welche Gene für das Überleben des Parasiten notwendig sind und wo die entsprechenden Genprodukte, d. h. Proteine, im Malariaerreger lokalisiert sind. Ihre Methode haben sie in der Fachzeitschrift Nature Methods veröffentlicht.
Dr. Tobias Spielmann, Arbeitsgruppenleiter am BNITM erklärt: „Unsere Methode nennt sich Selection Linked Integration (SLI). Damit konnten wir u. a. zum ersten Mal die besondere Bedeutung des Parasiten-Proteins Kelch13 aufzeigen, das verantwortlich gemacht wird für die Resistenz gegen Artemisinin, dem wichtigsten Malariamedikament.“
Das SLI-System ermöglicht es, an einer bestimmten Stelle im Genom der Parasiten neue Genabschnitte einzubringen, um so Gene auszuschalten, andere Gene einzubringen oder die entsprechenden Proteine mit einem Marker zu versehen. Neu dabei ist, dass die Parasiten mit veränderten Genomen gezielt selektioniert werden und das System mit vielen anderen Methoden kombinierbar ist. Traditionell wurden Veränderungen im Genom passiv selektioniert, was oft viele Monate in Anspruch nahm oder gar nicht zum Erfolg führte. Damit erweist sich die neue Methode als Alternative zum weit verbreiteten CRISPR/Cas System, welches für ähnliche genetische Veränderungen angewandt wird.
„Die größten Vorteile unseres SLI-Systems sind die Zeitersparnis und die Einfachheit, genetische Veränderung zu erhalten, sowie die Möglichkeit, damit überlebensnotwendige Gene bzw. Proteine auf vielerlei Weise zu untersuchen“, betont Spielmann. Dass die SLI-Technik wirklich robust funktioniert, konnte die AG Spielmann bereits durch die Untersuchungen von einer ganzen Reihe von Proteinen demonstrieren.
SLI-Technik ermöglicht funktionelle Analyse resistenter Parasiten
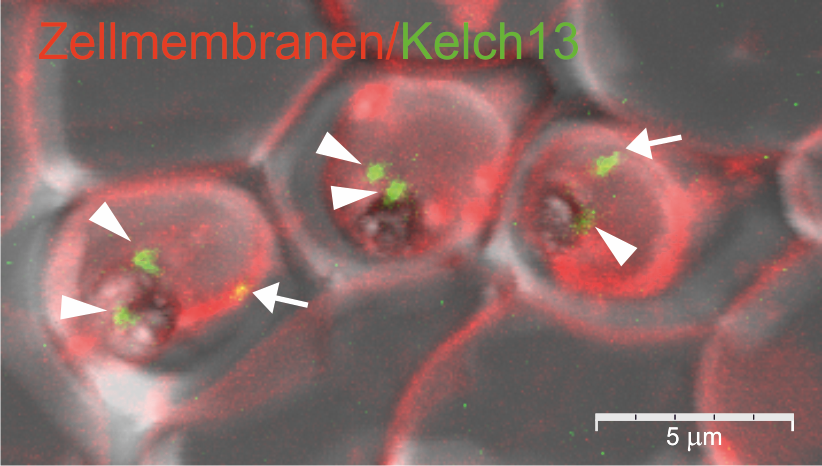
Obwohl die Artemisinin-Resistenz des Malariaerregers P. falciparum schon seit längerem auf eine Mutation im Gen kelch13 zurückgeführt wird, war bisher nicht bekannt, welche Rolle das Genprodukt Kelch13 in den Parasiten spielt. Um seine Funktion zu untersuchen, konnte die AG Spielmann mit Hilfe der SLI-Technik Kelch13 mit einem fluoreszierenden Markerprotein verknüpfen und zum ersten Mal seinen Aufenthaltsort in der Parasitenzelle sichtbar machen. Des Weiteren konnte die AG Spielmann durch die Kombination von SLI mit weiteren Verfahren erstmals zeigen, dass Kelch13 benötigt wird, damit sich die Parasiten in den roten Blutzellen vermehren können.
Zu zukünftigen Projekten der Arbeitsgruppe sagt Spielmann: „Unsere SLI-Technik wird die Malaria-Forschung beschleunigen. Wir können damit die unbekannte Biologie des Erregers analysieren. Wir erwarten, dass damit in kurzer Zeit neue Erkenntnisse zum Mechanismus der Artemisinin-Resistenz und zur Funktion anderer Parasiten-Proteinen möglich sind. Solche Erkenntnisse werden benötigt, um die Entwicklung neuer Malariamedikamente voranzutreiben.
Veröffentlichung:
Jakob Birnbaum, Sven Flemming, Nick Reichard, Alexandra Blancke Soares, Paolo Mesén-Ramírez, Ernst Jonscher, Bärbel Bergmann & Tobias Spielmann. A genetic system to study Plasmodium falciparum protein function; Nature Methods; Advance Online Publication 13 March 2017 http://dx.doi.org/10.1038/nmeth.4223
Über das BNITM:
Das Bernhard-Nocht-Institut für Tropenmedizin (BNITM) ist Deutschlands größte Einrichtung für Forschung, Versorgung und Lehre auf dem Gebiet tropentypischer Erkrankungen und neu auftretender Infektionskrankheiten. Aktuelle Forschungsschwerpunkte bilden Malaria, hämorrhagische Fieberviren, Tuberkulose und Gewebewürmer. Für den Umgang mit hochpathogenen Viren und infizierten Insekten verfügt das Institut über Laboratorien der höchsten biologischen Sicherheitsstufe (BSL4) und ein Sicherheits-Insektarium (BSL3). Das BNITM umfasst das nationale Referenzzentrum für den Nachweis aller tropischen Infektionserreger. Gemeinsam mit dem ghanaischen Gesundheitsministerium und der Universität von Kumasi, Ghana, betreibt es ein modernes Forschungs- und Ausbildungszentrum im westafrikanischen Regenwald, das auch externen Arbeitsgruppen zur Verfügung steht.
In Verbindung stehende Artikel:
Malaria parasite filmed in 3D - 02.03.2016 22:54
Malariaparasit in 3D gefilmt - 18.02.2014 12:07
Links:
http://dx.doi.org/10.1038/nmeth.4223
http://www.ndr.de/info/Malariaforschung-erzielt-weiteren-Fortschritt,audio318134.html


Dr. Eleonora Schönherr
Telefon: +49 40 285380-269
E-Mail: presse@bnitm.de
Ansprechperson
Dr. Tobias Spielmann
Research group leader
Telefon : +49 40 285380-486
E-Mail : spielmann@bnitm.de
Dr. Eleonora Schönherr
Presse- & Öffentlichkeitsarbeit
Telefon : +49 40 285380-269
E-Mail : presse@bnitm.de







